Differenza tra riparazione non conformità e riparazione di emissione nucleotidi | Riparazione di non conformità vs riparazione di excision di nucleotidi
Differenza chiave - Riparazione di mismatch vs riparazione di escissione di nucleotidi
Dieci e migliaia di danni del DNA si verificano nella cellula al giorno. Induce le modifiche ai processi cellulari come la replica, la trascrizione e la vitalità della cellula. In alcuni casi, le mutazioni causate da questi danni del DNA possono portare a malattie deleteri come i tumori e le sindromi associate all'invecchiamento (es.: Progeria). Indipendentemente da questi danni, la cellula inizia un meccanismo di riparazione a cascata altamente organizzato chiamato risposte al danno del DNA. Molti sistemi di riparazione del DNA sono stati identificati nel sistema cellulare; questi sono noti come riparazione di escissione di base (BER), riparazione non corrispondente (MMR), riparazione di escissione di nucleotidi (NER), riparazione di rottura a doppio filamento. La riparazione di escissione di nucleotidi è un sistema altamente versatile che riconosce le lesioni del DNA di distorsione ingombrante dell'elica e li rimuove. D'altra parte, la ripartizione non corrispondente sostituisce le basi misincorporated durante la replica. La differenza fondamentale tra la riparazione di mismatch e la riparazione di escissione dei nucleotidi è che la riparazione di escissione di nucleotidi (NER) viene utilizzata per rimuovere i dimeri pirimidina formati da irradiazione UV e lesioni ingombranti dell'elica causate da addotti chimici, mentre il sistema di riparazione non corrispondente svolge un ruolo importante nella correzione dei misincorporated basi che sono sfuggite agli enzimi di replicazione (DNA polimerasi 1) durante la postreplicazione. Oltre alle basi non corrispondenti, le proteine del sistema MMR possono anche riparare i loop di inserzioni / delezioni (IDL) che sono i risultati dello scivolamento della polimerasi durante la replicazione di sequenze di DNA ripetitivi.
SOMMARIO
1. Panoramica e differenza chiave
2. Che cosa è la riparazione non conforme
3. Che cosa è Nucleotide Excision Repair
4. Confronto Side By Side - Riparazione di non conformità rispetto alla riparazione di eliminazione dei nucleotidi
5. Riepilogo
Che cosa è la riparazione di Excision Nucleotide?
La caratteristica più importante della riparazione di escissione di nucleotidi è quella di riparare i danni modificati del nucleotide causati da significative distorsioni nella doppia elica del DNA. È osservato in quasi tutti gli organismi che sono stati esaminati fino ad oggi. Uvr A, Uvr B, Uvr C (eccinucleasi) Uvr D (a helicase) sono gli enzimi più noti coinvolti nel NER che innescano la riparazione del DNA nell'organismo modello Ecoli. Il complesso enzimatico multi-subunità di Uvr ABC produce i polipeptidi Uvr A, Uvr B, Uvr C.I geni codificati per i suddetti polipeptidi sono Uvr A, Uvr B, uvr C. Gli enzimi Uvr A e B riconoscono collettivamente la distorsione indotta da danni causata dall'elica doppia del DNA, come i dimmers di pirimidina dovuta all'irraggiamento UV. Uvr A è un enzima di ATPasi e questa è una reazione autocatalitica. Poi Uvr A lascia il DNA mentre il complesso Uvr BC (nucleasi attiva) colpisce il DNA in entrambi i lati del danno che catalizzato da ATP. Un'altra proteina chiamata Uvr D codificata dal gene uvrD è un enzima di elicasi II che rilascia il DNA che risulta dal rilascio di un segmento DNA danneggiato a singola flotta. Questo lascia un divario nell'elica del DNA. Dopo che il segmento danneggiato è stato tagliato, uno spazio di 12-13 nucleotidi rimane nel filo del DNA. Questo è riempito dall'enzima DNA polymerasi I e il nick è sigillato dalla ligasi del DNA. ATP è richiesto a tre fasi di questa reazione. Il meccanismo NER può essere identificato anche negli esseri umani come i mammiferi. Nell'uomo, la condizione cutanea chiamata Xeroderma pigmentosum è dovuta ai dimeri del DNA causati dall'irraggiamento UV. I geni XPA, XPB, XPC, XPD, XPE, XPF e XPG producono proteine per sostituire i danni del DNA. Le proteine dei geni XPA, XPC, XPE, XPF e XPG hanno l'attività della nucleasi. D'altra parte, le proteine dei geni XPB e XPD mostrano l'attività di elicasi che analoghi a Uvr D in E coli.

Figura 01: Riparazione di escissione di nucleotidi
Che cosa è la riparazione non corrispondente?
Il sistema di riparazione non corrispondente viene avviato durante la sintesi del DNA. Anche con la subunità funzionale, la DNA polimerasi III consente l'incorporazione di un nucleotide sbagliato per la sintesi ogni 10 8 coppie di base. Le proteine di riparazione non corrispondenti riconoscono questo nucleotide, l'accise e lo sostituiscono con il corretto nucleotide responsabile del grado finale di accuratezza. La metilazione del DNA è fondamentale per le proteine MMR per riconoscere il filamento genitore dal filamento appena sintetizzato. La metilazione del nucleotide di adenina (A) in un motivo GATC di un filo appena sintetizzato è un po 'ritardato. D'altro canto, il nucleotide adenino a filo genitore in motivo GATC è già metilato. Le proteine MMR riconoscono il filo appena sintetizzato da questa differenza dal filamento genitore e riparano la ripartizione di mismatch in un filo appena sintetizzato prima che venga metilato. Le proteine MMR dirigono la loro attività di riparazione ad accise il nucleotide sbagliato prima che il filo DNA di recente replicato viene metilato. Gli enzimi Mut H, Mut L e Mut S codificati da geni mut H, mut L, mut S catalizzano queste reazioni in Ecoli. La proteina Mut S riconosce sette su otto possibili coppie di base non corrispondenti ad eccezione di C: C e si lega al sito di mismatch nel DNA duplex. Con ATP legati, Mut L e Mut S entrano nel complesso in seguito. Il complesso trasloca qualche migliaio di coppie di base fino a trovare un motivo ematilato GATC. L'attività nuclease dormiente di Mut H proteina viene attivata una volta che trova un motivo ematilato GATC. Si fila il filo di DNA nonmetilato lasciando un nick 5 'a nucleotide G di motivo GATC non metallizzato (filamento di DNA recentemente sintetizzato).Poi lo stesso filo dall'altro lato della disallineazione viene intaccato da Mut H. Nel resto delle fasi, le azioni collettive di Uvr D una proteina di elicasi, Mut U, SSB ed esonuclease I accisero il nucleotide non corretto nel singolo filamento DNA. Il divario formato nell'emissione viene riempito dalla DNA polimerasi III e sigillata mediante ligasi. Un sistema simile può essere identificato nei topi e negli esseri umani. La mutazione di hMLH1 umano, hMSH1 e hMSH2 sono coinvolti nel cancro del colon ereditario nonpolyposis che deregola la divisione cellulare delle cellule del colon.
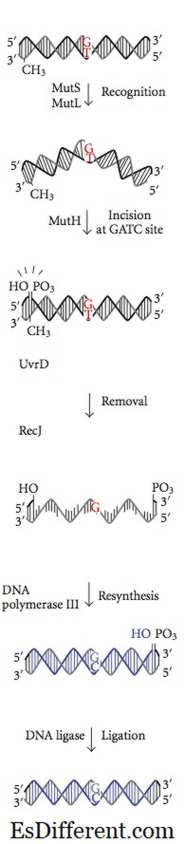
Figura 02: Riparazione non corrispondente
Qual è la differenza tra la riparazione non corrispondente e la riparazione dell'emissione nucleotidica?
- diff articolo prima della tabella ->
Riparazione di non corrispondenza rispetto alla riparazione di emissione di nucleotidi |
|
| Il sistema di riparazione non corrispondente si verifica durante la post replicazione. | Ciò riguarda la rimozione di dimeri di pirimidina a causa dell'irradiazione U. e di altre lesioni del DNA a causa dell'addotto chimico. |
| Gli enzimi | |
| E 'catalizzato da Mut S, Mut L, Mut H, Uvr D, SSB ed esonucleasi I. | E' catalizzato da enzimi Uvr A, Uvr B, Uvr C e UvrD. |
| Metilazione | |
| È fondamentale iniziare la reazione. | La metilazione del DNA non è necessaria per avviare la reazione. |
| Azione degli enzimi | |
| Mut H è una endonucleasi. | Uvr B e Uvr C sono esonucleasi. |
| Occasione | |
| Ciò avviene specificamente durante la replica. | Questo accade quando esposto a mutageni U. V o chimici, non durante la replica |
| Conservazione | |
| È altamente conservato | Non è altamente conservato. |
| Gap Filling | |
| Viene eseguito da DNA polymerase III. | E 'fatta dalla DNA polimerasi I. |
Riepilogo - Riparazione di mismatch vs riparazione di escissione di nucleotidi
La riparazione non corrispondente (MMR) e la riparazione di escissione del nucleotide (NER) sono due meccanismi che si verificano nella cella per rettificare Danni e distorsioni del DNA che sono causati da vari agenti. Questi sono chiamati collettivamente come meccanismi di riparazione del DNA. La riparazione di escissione del nucleotide ripara i danni nucleotidici modificati, tipicamente quei dannosi danni dell'elica doppia del DNA che si verificano a causa dell'esposizione ad irraggiamento U. V e adotti chimici. Le proteine di riparazione non corrispondenti riconoscono il nucleotide sbagliato, l'accisa e la sostituiscono con un nucleotide corretto. Questo processo è responsabile del grado finale di accuratezza durante la replica.
Riferimento:
1. Cooper, Geoffrey M. "Riparazione del DNA. "La cellula: un approccio molecolare. Seconda edizione. U. S. National Library of Medicine, 01 gennaio 1970. Web. 09 marzo 2017.
2. "Meccanismi e funzioni di riparazione di mismatch del DNA. "Ricerca cellulare. U. S. Biblioteca Nazionale di Medicina, n. d. Web. 09 Mar. 2017.
Immagine per gentile concessione:
1. "Nucleotide Excision Repair-journal. PBIO. 0040203. g001 "di Jill O. Fuss, Priscilla K. Cooper - (CC BY 2. 5) via Wikimedia Commedia
2. "Il DNA non corrisponde alla riparazione di Ecoli" di Kenji Fukui - (CC BY 4. 0) via Commons Wikimedia




